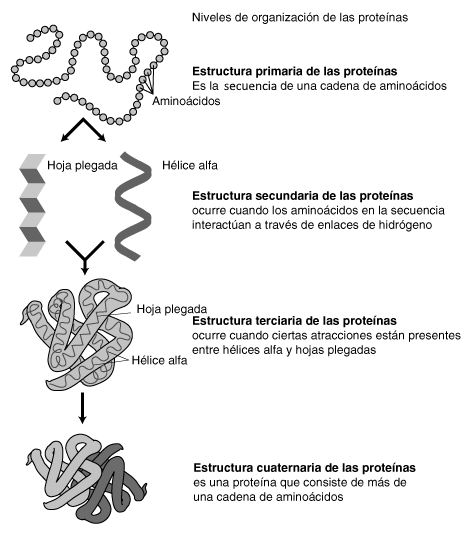
viene determinada por la secuencia de AA en la cadena proteica, es decir, el número de AA presentes y el orden en que están enlazados (Figura de la derecha). Las posibilidades de estructuración a nivel primario son prácticamente ilimitadas. Como en casi todas las proteínas existen 20 AA diferentes, el número de estructuras posibles viene dado por las variaciones con repetición de 20 elementos tomados de n en n, siendo n el número de AA que componen la molécula proteica.
Generalmente, el número de AA que forman una proteína oscila entre 80 y 300. Los enlaces que participan en la estructura primaria de una proteína son covalentes: son los enlaces peptídicos. El enlace peptídico (Figura de la izquierda) es un enlace amida que se forma entre el grupo carboxilo de una AA con el grupo amino de otro, con eliminación de una molécula de agua. Independientemente de la longitud de la cadena polipeptídica, siempre hay un extremo amino terminal y un extremo carboxilo terminal que permanecen intactos. Por convención, la secuencia de una proteína se lee siempre a partir de su extremo amino (Figura superior).
Como consecuencia del establecimiento de enlaces peptídicos entre los distintos AA que forman la proteína se origina una cadena principal o "esqueleto" a partir del cual emergen las cadenas laterales de los AA (Átomos sombreados en la Figura de la derecha).Los átomos que componen la cadena principal de la proteína son el N del grupo amino (condensado con el AA precedente), el Ca (a partir del cual emerge la cadena lateral) y el C del grupo carboxilo (que se condensa con el AA siguiente). Por lo tanto, la unidad repetitiva básica que aparece en la cadena principal de una proteína es: (-NH-Ca-CO-)
Como la estructura primaria es la que determina los niveles superiores de organización, el conocimiento de la secuencia de AA es del mayor interés para el estudio de la estructura y función de una proteína. Clásica mente, la secuenciación de una proteína se realiza mediante métodos químicos. El método más utilizado es el de Edman, que utiliza el fenilisotiocianato para marcar la proteína (representado en la Figura de la izquierda como un triángulo) e iniciar una serie de reacciones cíclicas que permiten identificar cada AA de la secuencia empezando por el extremo amino. Hoy en día esta serie de reacciones las realiza de forma automática un aparato llamado secuenciador de AA.
La estructura secundaria
de una proteína es la que adopta especialmente. Existen ciertas estructuras repetitivas encontradas en las proteínas que permiten clarificarlas en dos tipos: hélice alfa y lámina beta.
Una hélice alfa es una apretada hélice formada por una cadena polipeptídica. La cadena polipetídica principal forma la estructura central, y las cadenas laterales se extienden por fuera de la hélice. El grupo carboxílo (CO) de un aminoácido n se une por puente hidrógeno al grupo amino (NH) de otro aminoácido que está tres residuos mas allá ( n + 4 ). De esta manera cada grupo CO y NH de la estructura central (columna vertebral o "backbone") se encuentra unido por puente hidrógeno.
Existen tres modelos de alfa hélice. El primero muestra solo al carbono alfa de cada aminoácido. El segundo muestra todos los átomos que forman la columna vertebral del polipéptido .
El tercero y mas completo modelo, muestra todos los puentes hidrógeno que mantienen la alfa-hélice. Las hélices generalmente están formadas por aminoácidos hidrófobos, en razón que son, generalmente, la máxima atracción posible entre dichos aminoácidos. Las hélices se observan, en variada extensión, prácticamente en todas las proteínas.
B-Las láminas beta son el otro tipo de estructura secundaria. Pueden ser paralelas o antiparalelas. Las anti-paralelas generalmente se ven así:
Y los giros que tienen en su estructura :
donde el aminoácido n se une por puente hidrógeno al aminoácido (n +3) .
Existe un tipo especial de modelo molecular para resaltar la estructura secundaria de las proteínas. Este tipo de modelo de proteína representa los segmentos de lámina-beta como cintas en flecha (ribbons) y las alfa hélices como como cintas en espiral.
Prión en su forma PrPsc presenta gran proporción de láminas beta (43% láminas beta, 30% hélices alfa).
El resto de la cadena polipeptídica se referencia como un espiral al azar ("random coil"), y se dibuja como una fina línea. Por favor, note que espiral al azar o "random coil" es un nombre que lleva a confusión, dado que las proteínas están altamente organizadas, pero esta región no tiene una estructura secundaria con componentes difíciles de categorizar.
La estructura terciaria
es la estructura plegada y completa en tres dimensiones de la cadena polipeptídica, la hexoquinasa es una estructura tridimensional completa.
A diferencia de la estructura secundaria, la estructura terciaria de la mayor parte de las proteínas es específica de cada molécula, además, determina su función.
EL plegamiento terciario no es inmediato, primero se agrupan conjuntos de estructuras denominadas dominios que luego se articulan para formar la estructura terciaria definitiva. Este plegamiento está facilitado por uniones denominadas puentes disulfuro, -S-S- que se establecen entre los átomos de azufre del aminoácido cisteína.
Existen, sin embargo dos tipos de estructuras terciarias básicas:
- Proteínas fibrosas, insolubles en agua, como la alfa queratina o el colágeno.
- Proteínas globulares, solubles en agua.
La estructura cuaternaria
Solo está presente si hay mas de una cadena polipeptídica. Con varias cadenas polipeptídicas, la estructura cuaternaria representa su interconexión y organización. Esta es la imagen de la hemoglobina, una proteína con cuatro polipéptidos, dos alfa-globinas y dos beta globinas. En rojo se representa al grupo hem (complejo pegado a la proteína que contiene hierro, y sirve para transportar oxígeno).
No hay comentarios:
Publicar un comentario